扩增子分析|一行代码实现网络属性和节点计算及Zi-Pi绘制之R包ggClusterNet包进行微生物网络分析和绘制
一、引言
R包ggClusterNet由南京农业大学袁军教授和中科院刘永鑫团队开发,并发布在iMeta上。该工具广受微生物生态学研究者的喜爱和关注。其主要特点包括:快速分析、通过少量代码即可完成网络分析,且具备高度的可重复性。尤其是ggClusterNet提供了多种微生物生态网络可视化工具,支持对网络特性的深度挖掘。

图源:Wen,Tao, et al. 2022. iMeta 1, e32.
上期我们介绍ggClusterNet的绘图代码,重点展示其提供的多达十种网络布局选项,帮助研究者解决网络图布局不美观的问题,同时避免了专业软件Cytoscape和Gephi学习难度较高的障碍,需要的小伙伴可以通过扩增子分析|基于R语言ggClusterNet包进行生态网络分析1—十种可视化布局包括igraph,Gephi和maptree交流学习。我们还有一期关于R包micreco构建网络分析的内容,扩增子分析|基于R语言microeco包进行微生物群落网络分析(network网络、Zi-Pi关键可跳转至交流与学习。本期我们集中于网络特性深度挖掘。网络拓扑结构特性不仅能够揭示网络的复杂性,还能反映其稳定性,这也是网络分析中的重要指标。
二、网络特性指标解读
我们总结了周集中老师团队于2012年发表在BMC Bioinformatics和2021年发表在nature climate change上的内容,总结了一些网络特性指标。如下所示:
网络大小(Network Size)
网络的大小由节点的总数决定,较大的网络一般对扰动有更强的抵抗力。然而,网络的稳定性还受到其他拓扑指标的影响,网络大小本身并不直接暗示稳定性。
复杂性(Complexity)
复杂性通常与网络的多样性和连接强度相关联,复杂的网络能更好地抵御环境变化。一些研究表明,复杂性增加通常与稳定性增强相关联,但也有例外情况。
连接密度(Linkage Density)
链接密度是已实现边数与可能边数的比值,高链接密度可以增加网络的复杂性,从而提高其稳定性。复杂性增加的同时,某些研究也指出其可能降低网络的稳定性。
介数中心性(Betweenness Centrality)
介数中心性衡量节点在网络中作为“桥梁”的重要性,值越高表明节点对网络整体结构的贡献越大。高介数中心性的节点丧失可能导致网络更大程度的分裂。
脆弱性(Vulnerability)
脆弱性衡量某个节点对网络整体效率的贡献,当节点及其关联的边被移除时,脆弱性会反映效率的损失。低脆弱性通常意味着网络具有较高的稳定性。
关键物种(Keystone species)
关键物种是对网络结构和功能有重要影响的物种。研究表明,关键物种的存在与网络稳定性呈正相关。
平均路径长度(Average Path Length)
平均路径长度表示网络中节点间的平均最短路径,较短的路径长度通常表明资源共享效率较高。虽然短路径长度提高了效率,但也可能增加网络对局部扰动的脆弱性。
度(Degree)
节点度数衡量一个节点连接的边的数量,平均度数越高,网络越复杂。高度数的节点通常是网络中的枢纽,其增加通常与网络稳定性增强相关。
凝聚力(Cohesion)
凝聚力衡量网络中正负交互的平衡,高负向交互比例有助于网络稳定性。负向交互通过控制干扰的负面影响来增加网络的稳定性。凝聚力我们有一期内容进行详细解释并提供代码,可跳转至正负内聚力在R中的复现:跟着Nature系列文章学习如何评价群落中物种协作和竞争以及网络稳定性交流与学习。
模块化指数(Modularity)
模块化性衡量网络中不同子群或模块的分隔程度,高模块化性可以限制局部扰动扩散到整个网络。研究表明,高模块化性通常与网络的稳定性增强相关联。
聚类系数(Clustering Coefficient)
聚类系数衡量节点邻居之间的连接程度,较高的聚类系数通常表示网络的紧密性。高聚类系数虽然提高了局部稳定性,但可能使网络对全局扰动更加脆弱。
鲁棒性(Robustness)
鲁棒性指网络在受到干扰时维持其结构的能力。高鲁棒性意味着即使某些关键节点被移除,网络仍能维持其大部分功能。
连接性(Connectivity)
连接性指的是网络中节点之间形成的连接程度,高连接性提供了冗余,使网络对局部损失具有较强的恢复力。尽管有研究认为高连接性增加了系统的脆弱性,但总体认为高连接性可以提升稳定性。
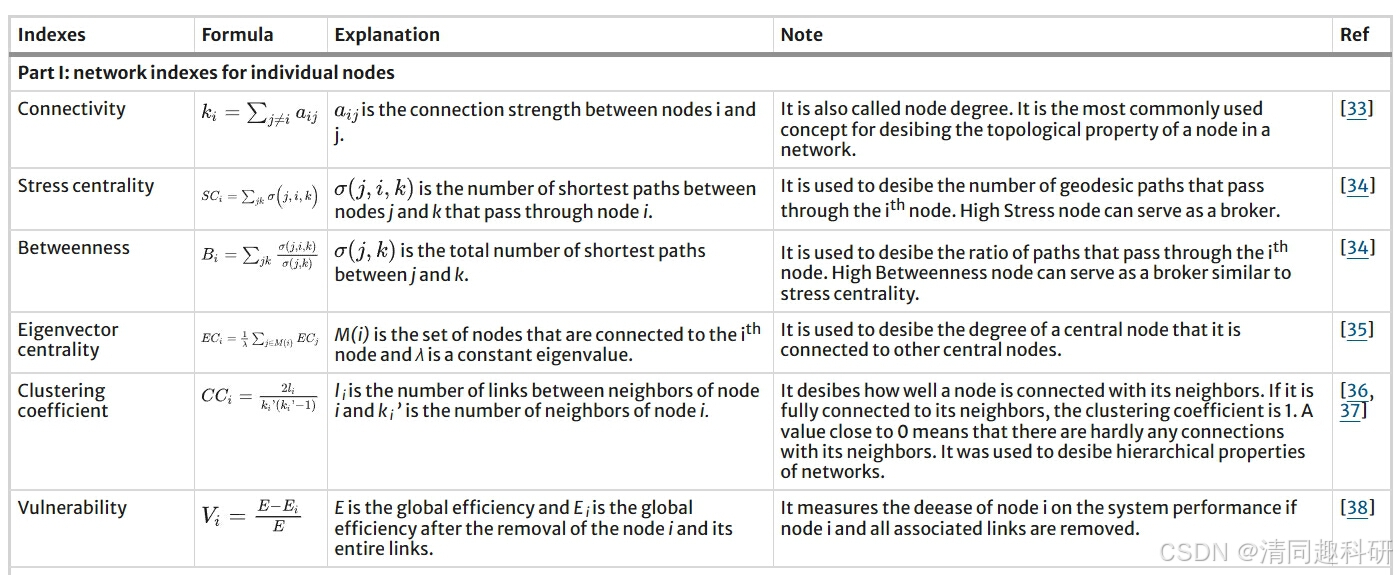
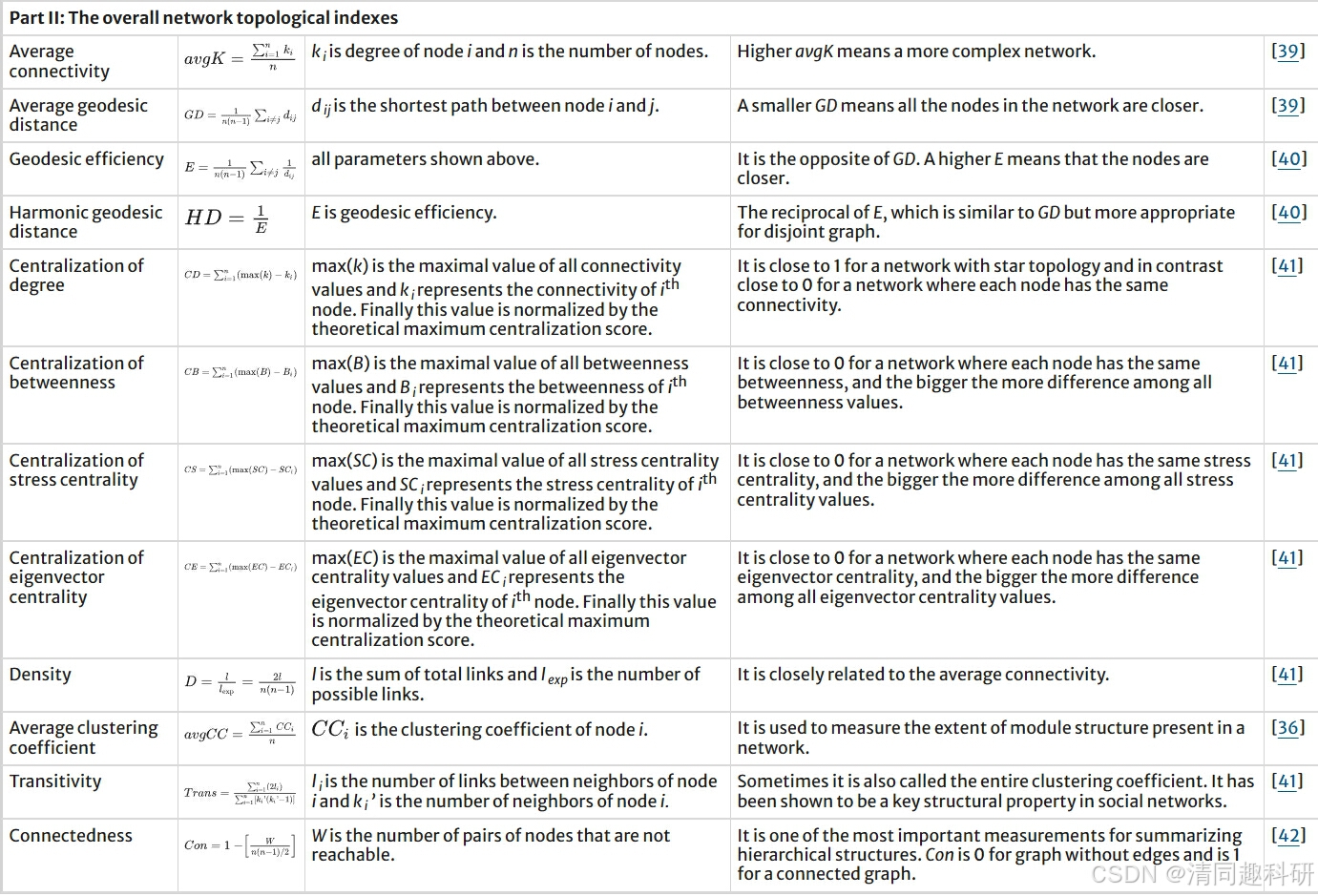
表格来源:Deng, Y., Jiang, YH., Yang, Y. et al. BMC Bioinformatics 13, 113 (2012).
三、示例数据和R代码
这套代码基于ggClusterNet包自带的数据进行分析,同时我们也提供了读取自己数据的方法,代码均可留言,小编会及时发送原始代码和示例数据。
🌟数据基本操作之包安装,微生物网络模型构建和分析数据准备
# 清理工作环境中的所有对象
rm(list = ls())
#检测并安装所需R包#
if (!require("phyloseq")) { install.packages("phyloseq") }
if (!require("igraph")) { install.packages("igraph") }
if (!require("network")) { install.packages("network") }
if (!require("sna")) { install.packages("sna") }
if (!require("tidyverse")) { install.packages("tidyverse") }
library(devtools)
if (!require("ggClusterNet")) {remotes::install_github("taowenmicro/ggClusterNet") }
if (!require("ggrepel")) { install.packages("ggrepel") }
if (!require("tidyfst")) { install.packages("tidyfst") }
#导入所需R包
library(phyloseq)
library(igraph)
library(network)
library(sna)
library(tidyverse)
library(ggClusterNet)
library(ggrepel)
#第一种数据读取方法—--####加载包里的数据###
data(ps)
#第二种数据读取方法———###读取自己输入的数据#
###使用的时候把代码前面的#删除即可,metadata,taxonomy,tree数据非必须###
#metadata = read.csv("metadata.csv",row.names = 1)
#otutab = read.csv("otu_table.csv", row.names=1)
#taxonomy = read.csv("tax_table.csv", row.names=1)
#tree = read_tree("otus.tree")
# 创建 phyloseq 对象
#ps = phyloseq(
sample_data(metadata),
otu_table(as.matrix(otutab), taxa_are_rows = TRUE),
tax_table(as.matrix(taxonomy)),
phy_tree(tree)
#)
###########计算微生物网络#################
result = corMicro (ps = ps,
N = 250,
r.threshold=0.8,
p.threshold=0.05,
method = "pearson"
)
#提取数据
cor = result[[1]]
dim(cor)
ps_net = result[[3]]
# nodeEdge 函数生成节点和边的文件
result4 = nodeEdge(cor = cor)
# 提取边的文件
edge = result4[[1]]
dim(edge) # 获取边文件的维度
edge$weight # 查看边的权重信息
# 提取节点文件
node = result4[[2]]
dim(node) # 获取节点文件的维度
# 将节点和边信息输入到 igraph 中,以便进行绘图
igraph = igraph::graph_from_data_frame(edge, directed = FALSE, vertices = node)
🌟 网络属性计算
#计算网络性质
dat = net_properties.2(igraph,n.hub = T)
dat
#将网络性质输出到netproperties.csv中,可供查阅
write.csv(dat,"netproperties.csv")
🌟 节点属性计算
#####################节点属性计算####################
#计算节点属性表
nodepro = node_properties(igraph)
dim(nodepro)
#将节点属性表写入到nodeproperties.csv表格中
write.csv(nodepro,"nodeproperties.csv")
🌟 合并节点属性和物种和注释信息
###########合并节点属性和物种和注释信息##########
nodeG = merge(nodes,nodepro,by = "row.names",all =FALSE)
dim(nodeG)
head(nodeG)
write.csv(nodeG,"nodeproperties1.csv")
pnet <- ggplot() + geom_segment(aes(x = X1, y = Y1, xend = X2, yend = Y2,color = as.factor(wei_label)),
data = edge, size = 0.5) +
geom_point(aes(X1, X2,fill = Phylum,size = igraph.degree),pch = 21, data = nodeG) +
scale_colour_brewer(palette = "Set1") +
scale_x_continuous(breaks = NULL) + scale_y_continuous(breaks = NULL) +
# labs( title = paste(layout,"network",sep = "_"))+
# geom_text_repel(aes(X1, X2,label=Phylum),size=4, data = plotcord)+
# discard default grid + titles in ggplot2
theme(panel.background = element_blank()) +
# theme(legend.position = "none") +
theme(axis.title.x = element_blank(), axis.title.y = element_blank()) +
theme(legend.background = element_rect(colour = NA)) +
theme(panel.background = element_rect(fill = "white", colour = NA)) +
theme(panel.grid.minor = element_blank(), panel.grid.major = element_blank())
pnet
🌟Zi-Pi图一键绘制
res = ZiPiPlot(igraph = igraph,method = "cluster_fast_greedy")
p <- res[[1]]
p
ggsave("1.png",p)
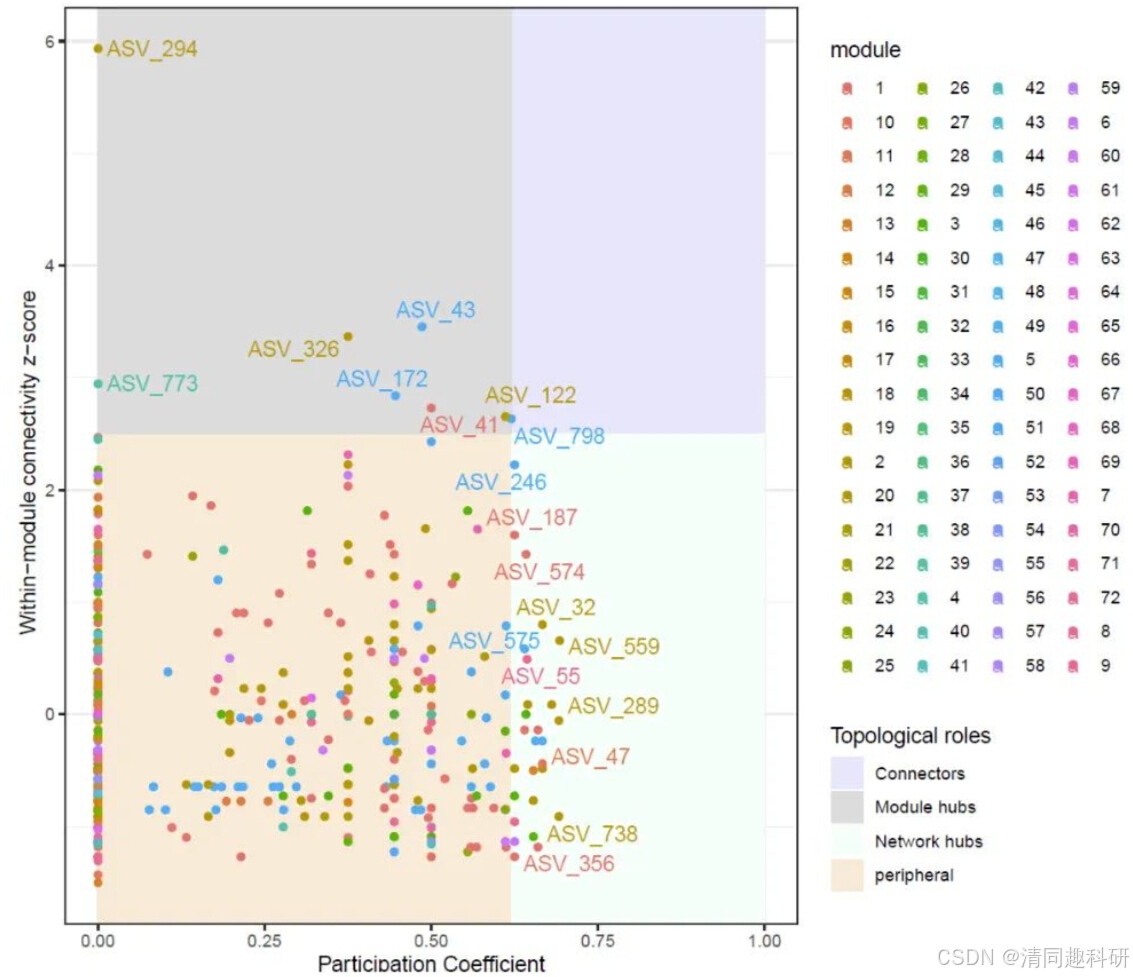
🌟Zi-Pi绘制详细代码
###########以下是zi-pi详细代码可酌情考虑#################
# 选择聚类方法,这里是 "cluster_fast_greedy"
fc <- igraph::cluster_fast_greedy(igraph, weights = abs(igraph::E(igraph)$weight))
# 计算模块化程度
modularity <- igraph::modularity(igraph, igraph::membership(fc))
# 按照模块为节点配色
comps <- igraph::membership(fc)
igraph::V(igraph)$module <- as.character(comps)
# 获取节点角色信息
taxa.roles <- module.roles(igraph)
# 处理标签,直接将 row.names 作为标签
taxa.roles$label <- row.names(taxa.roles)
# 过滤 z 和 p 值以确定哪些标签要显示
taxa.roles$label <- ifelse(taxa.roles$z > 2.5 | taxa.roles$p > 0.62, taxa.roles$label, "")
# 定义角色类型,根据 z 和 p 值分类
taxa.roles$role_7 <- taxa.roles$roles
taxa.roles[which(taxa.roles$z < 2.5 & taxa.roles$p < 0.62), 'roles'] <- 'Peripherals'
taxa.roles[which(taxa.roles$z < 2.5 & taxa.roles$p >= 0.62), 'roles'] <- 'Connectors'
taxa.roles[which(taxa.roles$z >= 2.5 & taxa.roles$p < 0.62), 'roles'] <- 'Module hubs'
taxa.roles[which(taxa.roles$z >= 2.5 & taxa.roles$p >= 0.62), 'roles'] <- 'Network hubs'
# 去掉 NA 值
taxa.roles <- na.omit(taxa.roles)
# 绘制角色图,使用 ggrepel 包来添加标签
p <- plot_roles2(taxa.roles) +
ggrepel::geom_text_repel(data = taxa.roles,
aes(x = p, y = z, color = module, label = label),
size = 4)
# 显示图
print(p)
🌟关键物种筛选
####################关键物种筛选###################
hub = hub_score(igraph)$vector %>%
sort(decreasing = TRUE) %>%
head(5) %>%
as.data.frame()
colnames(hub) = "hub_sca"
ggplot(hub) +
geom_bar(aes(x = hub_sca,y = reorder(row.names(hub),hub_sca)),stat = "identity",fill = "#4DAF4A")
四、参考文献
[1] Wen,Tao, Penghao Xie, Shengdie Yang, Guoqing Niu, Xiaoyu Liu, Zhexu Ding, Chao Xue, Yong-Xin Liu, Qirong Shen, and Jun Yuan. 2022. “ ggClusterNet: An R package for microbiome network analysis and modularity-based multiple network layouts.” iMeta 1, e32.
[2] Deng, Y., Jiang, YH., Yang, Y. et al. Molecular ecological network analyses. BMC Bioinformatics 13, 113 (2012).
[3] Yuan, M.M., Guo, X., Wu, L. et al. Climate warming enhances microbial network complexity and stability. Nat. Clim. Chang. 11, 343–348 (2021).
五、相关信息
!!!本文内容由小编总结互联网和文献内容总结整理,如若侵权,联系立即删除!
!!!有需要的小伙伴评论区获取今天的测试代码和实例数据。
📌示例代码中提供了数据和代码,小编已经测试,可直接运行。
以上就是本节所有内容。
如果这篇文章对您有用,请帮忙一键三连(点赞、收藏、评论、分享),让该文章帮助到更多的小伙伴。
本文地址:https://www.vps345.com/7102.html